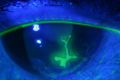
Биологи под руководством известного генетика Джорджа Черча создали метод секвенирования РНК in situ, который позволяет составить карту экспрессии тысяч генов в масштабах от субклеточного уровня до целого мозга или эмбриона. Описание метода опубликовано в Science, кратко о нем можно посмотреть в ролике Nature News.
Метод получил название FISSEQ (флюоресцентное секвенирование РНК in situ). Латинским выражением in situ биологи обозначают те методы, которые дают пространственную информацию, — данные которых привязаны к конкретной точке клетки или организма. В данном случае секвенирование FISSEQ позволяет определить последовательности молекул РНК, которые находятся в разных частях клетки. Секвенирование проходит с высоким, напоминающим оптическую микроскопию разрешением.
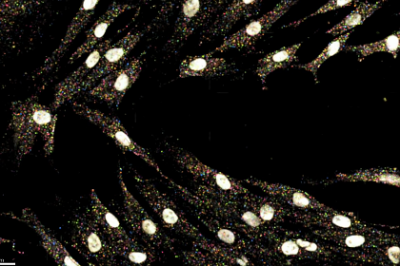
Процедура выглядит следующим образом. Сначала образец фиксируют и разрезают на тонкие слои. Затем из клеток удаляют мембраны, при этом РНК, ДНК и белки остаются фиксированы в матрице (этот метод «просветления» был изобретен недавно с целью получения прозрачного мозга). РНК с помощью обратной транскриптазы переводят в соответствующую ДНК-копию, которую затем многократно амплифицируют. В результате из одной молекулы РНК образуется кластер идентичных фиксированных молекул («наношарик»). В нем представлена не вся исходная РНК, а только ее фрагмент, но этого фрагмента достаточно для надежной идентификации всей молекулы. Затем используются уже известные методы пиросеквенирования: к образцу добавляют меченые основания, и во время синтеза новых копий ДНК в разных точках образца загораются разные сигналы.
До изобретения FISSEQ биологи могли либо установить пространственное распределение нескольких РНК (метод мечения in situ FISH), либо узнать последовательность всех РНК, но потерять при этом пространственную информацию (секвенирование). Новый метод сочетает обе возможности, что, вероятно, приведет к появлению нового класса исследований в эмбриологии и исследованиях мозга.